ESmol -- molecular viewer
ESmol - Visor Molecular para Android
¡DARSE CUENTA!
Si el dispositivo puede funcionar NDKmol, por favor utilice la misma. Es mucho más rápido y
tienen más características que ESmol.
Puede obtener NDKmol de https://play.google.com/store/apps/details?id=jp.sfjp.webglmol.NDKmol.
ESmol se mantiene por compatibilidad con dispositivos muy antiguos.
== == Sobre
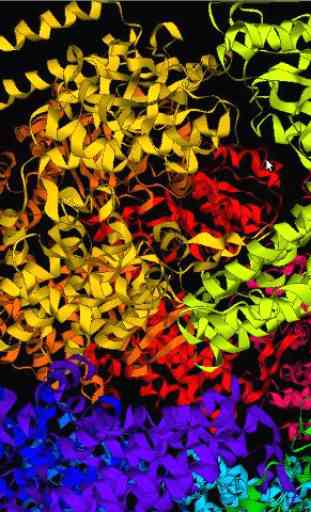
ESmol es un visor molecular para Android.
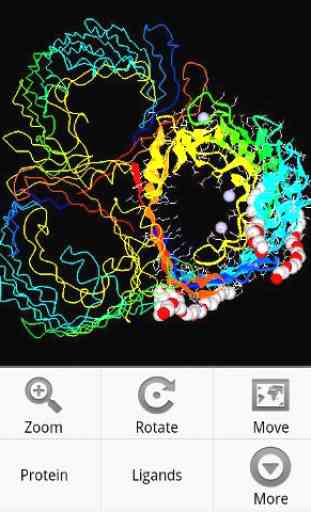
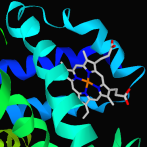
Puede ver estructuras tridimensionales de las proteínas, ácidos nucleicos y moléculas pequeñas. ESmol soporta la mayoría de representaciones comunes para las moléculas, tales como cinta, vestigios, palo, esfera y la línea. ESmol también soporta operaciones de simetría; conjuntos biológicos y cristal de embalaje se pueden visualizar. Puede buscar y descargar estructuras de RCSB AP y NCBI PubChem.
ESmol tiene casi la misma funcionalidad que GLmol, que está escrito en
WebGL / Javascript y se ejecuta en los navegadores web. Usted puede tratar de GLmol en http://webglmol.sourceforge.jp/index-en.html
== Características ==
* Leer archivo PDB
(ESmol no puede abrir moléculas de gran tamaño (más de 3 MB). Para ello, por favor utilice NDKmol en http://market.android.com/details?id=jp.sfjp.webglmol.NDKmol)
* Leer SDF archivo / MOL
* buscar y descargar estructuras de RCSB AP y NCBI PubChem
* Rotación / traslación / modelo de zoom por el dedo
* Representaciones
- Línea
- palo
- Esfera (van der Waals radio)
- huella de carbono alfa
- cinta
- Strand
- tubo de factor B
- escala de ácido nucleico
- La línea de ácido nucleico
- solvente 'estrellas'
* colorear
- Por cadena
- Por la estructura secundaria (cuando se define en registros HOJA / hélice)
- por elementos
- Gradación (a.k.a chainbow)
- factor B
- polar / no polar
* Cristalografía
- Unidad de visualización celular
- Mostrar cristal de embalaje (cuando se define en la sección OBSERVACIÓN)
- Visualización de montaje biológica (cuando se define en la sección OBSERVACIÓN)
== Cómo utilizar ==
Cuando se inicia, ESmol carga automáticamente desoxihemoglobina (PDBID: 4HHB) como un ejemplo. Puede girar la molécula por el dedo.
Para ampliar o traducir el botón molécula, pulse MENU en el teléfono / tableta y seleccionar el modo. También se admiten gestos con dos dedos.
Para cargar otros archivos PDB, por favor poner el archivo en el directorio "APP" de la tarjeta SD y seleccione "Abrir" en el MENÚ. También puede descargar directamente de estructuras RCSB AP y el servidor web NCBI PubChem. Seleccione "buscar y descargar" en el menú.
== == Contacto
página web del proyecto se encuentra en http://webglmol.sourceforge.jp/. También puede obtener los códigos fuente.
Comentarios y sugerencias son bienvenidos en http://sourceforge.jp/projects/webglmol/forums/ o [email protected]
=== === nota de licencia
ESmol en sí es bajo licencia GNU Licencia Pública General Reducida de la siguiente manera.
Sin embargo, los archivos PDB incluyen como ejemplos están en condiciones diferentes.
Por favor consulta
http://www.rcsb.org/pdb/static.do?p=general_information/about_pdb/pdb_advisory.html
----
ESmol - Visor Molecular para Android
(C) Copyright 2011, biochem_fan
Este programa es software libre: usted puede redistribuirlo y / o modificarlo
bajo los términos de la GNU Lesser General Public License publicada por
la Free Software Foundation, bien de la versión 3 de la Licencia, o
(A su elección) cualquier versión posterior.
Este programa se distribuye con la esperanza de que sea útil,
pero sin ninguna garantía; incluso sin la garantía implícita de
COMERCIALIZACIÓN o IDONEIDAD PARA UN PROPÓSITO PARTICULAR. ver el
GNU Lesser General Public License para más detalles.
Debería haber recibido una copia de la GNU Lesser General Public License
junto con este programa. Si no es así, consulte .
¡DARSE CUENTA!
Si el dispositivo puede funcionar NDKmol, por favor utilice la misma. Es mucho más rápido y
tienen más características que ESmol.
Puede obtener NDKmol de https://play.google.com/store/apps/details?id=jp.sfjp.webglmol.NDKmol.
ESmol se mantiene por compatibilidad con dispositivos muy antiguos.
== == Sobre
ESmol es un visor molecular para Android.
Puede ver estructuras tridimensionales de las proteínas, ácidos nucleicos y moléculas pequeñas. ESmol soporta la mayoría de representaciones comunes para las moléculas, tales como cinta, vestigios, palo, esfera y la línea. ESmol también soporta operaciones de simetría; conjuntos biológicos y cristal de embalaje se pueden visualizar. Puede buscar y descargar estructuras de RCSB AP y NCBI PubChem.
ESmol tiene casi la misma funcionalidad que GLmol, que está escrito en
WebGL / Javascript y se ejecuta en los navegadores web. Usted puede tratar de GLmol en http://webglmol.sourceforge.jp/index-en.html
== Características ==
* Leer archivo PDB
(ESmol no puede abrir moléculas de gran tamaño (más de 3 MB). Para ello, por favor utilice NDKmol en http://market.android.com/details?id=jp.sfjp.webglmol.NDKmol)
* Leer SDF archivo / MOL
* buscar y descargar estructuras de RCSB AP y NCBI PubChem
* Rotación / traslación / modelo de zoom por el dedo
* Representaciones
- Línea
- palo
- Esfera (van der Waals radio)
- huella de carbono alfa
- cinta
- Strand
- tubo de factor B
- escala de ácido nucleico
- La línea de ácido nucleico
- solvente 'estrellas'
* colorear
- Por cadena
- Por la estructura secundaria (cuando se define en registros HOJA / hélice)
- por elementos
- Gradación (a.k.a chainbow)
- factor B
- polar / no polar
* Cristalografía
- Unidad de visualización celular
- Mostrar cristal de embalaje (cuando se define en la sección OBSERVACIÓN)
- Visualización de montaje biológica (cuando se define en la sección OBSERVACIÓN)
== Cómo utilizar ==
Cuando se inicia, ESmol carga automáticamente desoxihemoglobina (PDBID: 4HHB) como un ejemplo. Puede girar la molécula por el dedo.
Para ampliar o traducir el botón molécula, pulse MENU en el teléfono / tableta y seleccionar el modo. También se admiten gestos con dos dedos.
Para cargar otros archivos PDB, por favor poner el archivo en el directorio "APP" de la tarjeta SD y seleccione "Abrir" en el MENÚ. También puede descargar directamente de estructuras RCSB AP y el servidor web NCBI PubChem. Seleccione "buscar y descargar" en el menú.
== == Contacto
página web del proyecto se encuentra en http://webglmol.sourceforge.jp/. También puede obtener los códigos fuente.
Comentarios y sugerencias son bienvenidos en http://sourceforge.jp/projects/webglmol/forums/ o [email protected]
=== === nota de licencia
ESmol en sí es bajo licencia GNU Licencia Pública General Reducida de la siguiente manera.
Sin embargo, los archivos PDB incluyen como ejemplos están en condiciones diferentes.
Por favor consulta
http://www.rcsb.org/pdb/static.do?p=general_information/about_pdb/pdb_advisory.html
----
ESmol - Visor Molecular para Android
(C) Copyright 2011, biochem_fan
Este programa es software libre: usted puede redistribuirlo y / o modificarlo
bajo los términos de la GNU Lesser General Public License publicada por
la Free Software Foundation, bien de la versión 3 de la Licencia, o
(A su elección) cualquier versión posterior.
Este programa se distribuye con la esperanza de que sea útil,
pero sin ninguna garantía; incluso sin la garantía implícita de
COMERCIALIZACIÓN o IDONEIDAD PARA UN PROPÓSITO PARTICULAR. ver el
GNU Lesser General Public License para más detalles.
Debería haber recibido una copia de la GNU Lesser General Public License
junto con este programa. Si no es así, consulte .
Categoría : Educación

Búsquedas relacionadas